Im Projekt «Doorstep» arbeiten die Forscher an Polymerpartikeln, die einen fluoreszierenden Farbstoff umgeben. Zersetzt die bakterielle Urease das Polymer, kann der Farbstoff seine Leuchtkraft entfalten.
Die Diagnostikmethode soll mit einem Rachenabstrich oder einer Sputum-Probe funktionieren. Dies würde es ermöglichen, die Erreger einer Lungenentzündung innert weniger Stunden anstelle von mehreren Tagen zu bestimmen.
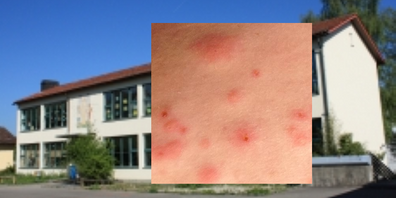
Pflaster warnt vor Wundkeimen
Ein wichtiges Anwendungsgebiet für eine schnelle und präzise Diagnose von resistenten Erregern sind zudem infizierte Wunden. Sie verursachen nicht nur Schmerzen und Gewebeschäden – sie sind auch eine Brutstätte für antibiotikaresistente Superkeime.
Ein Team um die Empa-Forscher Luciano Boesel und Giorgia Giovannini startet jetzt gemeinsam mit dem Kantonsspital St.Gallen ein Projekt, in dem sie einen Multisensorverband für Wunden entwickeln möchten.
Er basiert auf Silica-Nanopartikeln, die in einem widerstandsfähigen Hydrogel aus bioverträglichen Polymeren eingelagert sind. Die Sensortechnologie soll hierbei direkt in das Verbandmaterial integriert werden. Funktionalisiert werden die Nanopartikel mit Substanzen, die Ausscheidungen von bestimmten Bakterien spezifisch anzeigen können.
Sensoren reagieren auf Wundkeime
So sollen die Sensoren auf besonders gefürchtete Wundkeime wie Staphylococcus aureus reagieren und eine Veränderung des Säure-Base-Gleichgewichts in der Wunde anzeigen. Zudem soll das Risiko einer Antibiotikaresistenz rasch sichtbar werden.
Da hochpathogene Wundkeime über das Enzym Beta-Lactamase verfügen, mit dem sie bestimmte Antibiotika inaktivieren, enthält der Sensor Farbstoffe, die durch dieses Enzym gespaltet werden. Produzieren resistente Bakterien in der Wunde das Enzym, warnt der Sensor durch ein deutliches Leuchten unter UV-Licht.
Im Klinikalltag erlaubt der Wundsensor so eine schnelle kostengünstige Diagnose und eine personalisierte Wundbehandlung. Das Projekt wurde dank den grosszügigen Zuwendungen der Philipp und Henny Bender Stiftung, der Blumenau-Léonie Hartmann-Stiftung, der Hans Groeber-Stiftung sowie der Räschle Stiftung ermöglicht.
Aus dem Urin gefischt
Ein weiterer unangenehmer Vertreter aus dem Bakterienreich ist Pseudomonas aeruginosa. Das Stäbchenbakterium kann diverse Krankheiten hervorrufen, darunter Infektionen des Harntrakts etwa über Harnkatheter während eines Spitalaufenthalts. Und auch diese Erreger sind häufig resistent gegen diverse Antibiotika.
Ein Team aus Forschern der Empa und der ETH Zürich hat daher ein Verfahren mit magnetischen Nanopartikeln entwickelt, das die Bakterien schnell und präzise nachweist.
Da die Magnetpartikel an Eiweissbausteine gekoppelt sind, die ausschliesslich mit Pseudomonas aeruginosa reagieren, können die Bakterienzellen schliesslich über ein Magnetfeld spezifisch aus dem Urin «gefischt» werden.
Empfindlichkeit des Erregers analysieren
In einem nächsten Schritt wird die Empfindlichkeit der Erreger auf verschiedene Antibiotika mit einem Chemilumineszenz-Verfahren analysiert. Sind resistente Bakterien im Reagenzglas, strahlt die Probe Licht ab. Lassen sich die Keime hingegen mit Antibiotika abtöten, bleibt es dunkel.
«Alles in allem dauert der Resistenztest rund 30 Minuten – im Vergleich zu mehreren Tagen bei einer klassischen Anzucht von Bakterienkulturen», so Qun Ren, Gruppenleiterin am «Biointerfaces»-Labor der Empa in St.Gallen. So lässt sich innert Kürze die passende Antibiotika-Therapie ermitteln – und dadurch die Entstehung weiterer Resistenzen verhindern.